台灣農業研究
作者:林思妤、王昭月、陳涵葳、李承彬、林大鈞、杜元凱*
摘要:
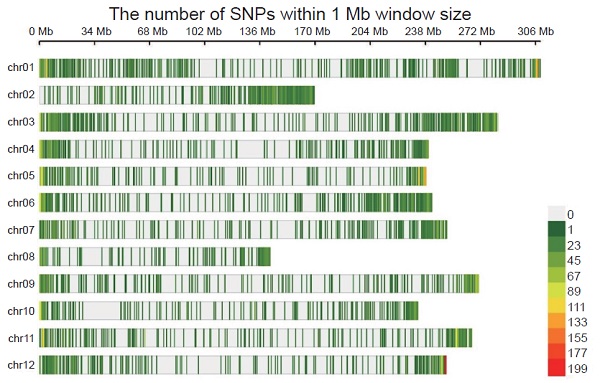
次世代定序技術已成為作物基因體研究相當常見的工具,而為集中定序資源與降低定序成本,已逐漸發展出多種簡化基因體定序技術,然而不同作物、不同族群組成所適宜的文庫建置方法 (如限制酶選擇或是定序量等) 有差異,若能針對目標作物及族群組成進行方法上的調整,將可使定序資源發揮最大效率。本研究使用簡化基因體技術之一:雙重酶切DNA 定序技術 (double-digest restriction site-associated DNA sequencing; ddRAD-seq) 針對不同種番椒 (Capsicum spp.) 種原基因體進行分析,第一部分比較PstI/MspI 及PstI/MseI 限制酶酵素組合,電腦模擬結果顯示兩種組合產生的可被定序片段為總片段之2.01% 及0.34%,而實際2 個酵素組合的定序結果顯示,以PstI/MspI 組合 (19–28 M) 比PstI/MseI 組合 (15–17 M) 得到較多成對且比對專一的讀序,因此不論在建庫資源利用上或是成功對應回參考序列的讀序數量/比例上,PstI/MspI 皆較PstI/MseI 表現佳。本研究第二部分針對28 個番椒收集系 (涵蓋5個番椒馴化種) 以PstI/MspI 酶切並進行ddRAD 定序,共得到14,230 個高品質單一核苷酸多型性 (single nucleotide polymorphism; SNP),平均分布於番椒12 對染色體並以染色體兩端基因較密集處較多;利用此些SNP 進行集群分析,亦能成功將不同種番椒品系正確分類。此說明本研究使用的一系列建庫、定序、生物資訊分析等方法,可用於橫跨不同種的番椒基因體分析,未來將以此為基礎,針對台灣番椒種原建立其基因體資訊,作為台灣番椒育種的根基。
關鍵字:簡化基因體定序、雙重限制酶、番椒、基因體分析
下載:![]() PDF 連結
PDF 連結